Introducción
Las poblaciones humanas de diferente ancestría y procedencia geográfica han mostrado diferente prevalencia de fenotipos complejos, como los relacionados con ciertas patologías o en la respuesta a determinados fármacos (Kreek, Bart, Lilly, Laforge y Nielsen, 2005). En particular, para la población argentina se han informado diferencias significativas en la variación de genes implicados en mecanismos de transmisión de la sensación dolorosa y la respuesta a analgésicos (López Soto y Catanesi, 2015; Raggio, González, Hohl, Glesmann y Catanesi, 2018). Entre los genes que influyen sobre estos mecanismos, el gen OPRM1 codifica el receptor opioide mu, el más importante del sistema opioide endógeno, el cual es activado principalmente por encefalinas y betaendorfinas del mismo sistema, y el que presenta mayor afinidad por la morfina como opioide exógeno. La activación de estos receptores reduce la sensación dolorosa y genera alivio. El gen OPRM1 presenta numerosos polimorfismos de nucleótido simple (SNPs), algunos de los cuales generan cambios en la estructura proteica, modificando la percepción del dolor y el efecto antinociceptivo de los fármacos opiáceos (López Soto, Agosti, Catanesi y Raingo, 2013; Owusu Obeng, Hamadeh y Smith, 2017; Palada, Kaunisto y Kalso, 2018).
Por otra parte, el gen COMT codifica la enzima catecol-O-metiltransferasa, que controla la neurotransmisión adrenérgica y dopaminérgica. Esta enzima cataliza la transferencia de un grupo metilo a uno de los grupos hidroxilo de las catecolaminas, en presencia de magnesio, para degradarlas. En mamíferos, es la principal enzima involucrada en la degradación metabólica de la dopamina liberada en la corteza frontal (Huerta et al., 2007). El gen COMT contiene un número considerable de SNPs, entre los cuales rs4680 es el más importante y ocasiona un cambio de aminoácido en la proteína codificada y en la modulación de la actividad dopaminérgica (Egan et al., 2001; Kim, Lee, Rowan, Brahim y Dionne, 2006; Henker et al., 2013; Palada et al., 2018).
Actualmente se está estudiando la prevalencia de variantes genéticas en los genes OPRM1 y COMT en poblaciones procedentes de diferentes zonas geográficas del mundo, y su posible relación con los fenotipos de percepción del dolor y respuesta a la analgesia (Rahim-Williams, Riley, Williams y Fillingim, 2012; Perry et al., 2019).
La provincia de Chaco, situada en el noreste de Argentina, posee una importante diversidad poblacional caracterizada por la coexistencia de comunidades tanto de origen nativo como foráneo. Presenta una elevada proporción de habitantes con ancestría nativa americana, a la que se sumó en las centurias recientes el ingreso de inmigrantes de origen principalmente europeo, pero también provenientes de otros países sudamericanos. Sobre esta base se formó la población criolla de origen mixto, a partir de inmigrantes y nativos chaqueños (Censo Nacional de Población, Hogares y Viviendas, 2010). En la actualidad, las comunidades de origen mixto habitan generalmente las zonas urbanas, mientras que algunos grupos nativos propios de la región chaqueña, conservan sus hábitos seminómades y pueden habitar tanto zonas urbanas como rurales (Martínez Sarasola, 2005; Maeder, 2012).
La diversidad genética de algunas poblaciones humanas de la provincia de Chaco ha sido estudiada utilizando marcadores genéticos no codificantes, principalmente de herencia uniparental, como son los que conforman haplotipos mitocondriales y de cromosoma Y (Bianchi et al., 1998; Demarchi y Mitchel, 2004; García y Demarchi, 2006; Bailliet et al., 2009; Di Fabio Rocca et al., 2015; Jurado Medina et al., 2021, entre otros), como así también marcadores genéticos de herencia biparental (Sala, Penacino y Corach, 1997, 1998; Tourret, López-Camelo y Vidal-Rioja, 1999; Tourret, Catanesi y Vidal-Rioja, 2000; Catanesi, Martina, Giovambattista, Zukas y Vidal-Rioja, 2007; Crosetti et al., 2008; Avena et al., 2012; Glesmann, Martina y Catanesi, 2013; Demarchi, 2014, entre otros). Por el contrario, las variaciones localizadas en regiones codificantes del ADN, que pueden incidir en la variación fenotípica, se han explorado en menor extensión (Dejean, Crouau-Roy, Goicoechea, Avena y Carnese, 2004; Parolin y Carnese, 2009; Catanesi y Glesmann, 2015; Raggio et al., 2018).
La población de Misión Nueva Pompeya (MNP) está ubicada en el noroeste de la provincia de Chaco, en el área fitogeográfica del Impenetrable. Esta localidad está expuesta a condiciones climáticas muy extremas y presenta escasas vías de comunicación hacia los principales centros urbanos (Altamirano y Salvay, 1996). La población de Misión Nueva Pompeya está compuesta actualmente por nativos de la comunidad wichí, criollos y residentes temporarios (empleados de la administración pública). Las comunidades wichí actuales proceden de los matacos de Salta, y mantienen con ellos estrechos lazos hasta el presente. Los wichí se encuentran dentro del grupo lingüístico mataco-mataguayo, los cuales, junto con los mataguayo, chorote y chulupí habitaban el Gran Chaco central y austral (Martínez Sarasola, 2005). Estas comunidades habitan zonas rurales y periurbanas en el oeste de la provincia de Formosa, el noroeste de Chaco, el este de Salta y el sudeste de Jujuy. De hábitos cazadores, recolectores y pescadores, actualmente muchos continúan con estas prácticas (Solá, 2009).
Dada la diversidad poblacional existente en la provincia de Chaco, es de particular interés analizar la variabilidad genética que poseen las poblaciones chaqueñas en regiones codificantes del genoma. En este trabajo se presenta la variación de los genes OPRM1 y COMT a través del análisis de marcadores genéticos de un nucleótido (SNPs) en individuos pertenecientes a la población de Misión Nueva Pompeya, y se compara la información obtenida con datos de la población de Resistencia y de la comunidad wichí de la provincia de Chaco (Raggio et al., 2018). El conocimiento de dicha información relacionada con la percepción del dolor es de interés no solo antropológico, sino también médico, teniendo en cuenta que en el futuro puede ser de utilidad para la toma de decisiones referidas al tratamiento y prevención del dolor en cada paciente (Pozo-Rosich, 2012). En este sentido, la medicina genómica personalizada depende del conocimiento de las variantes genéticas locales, para orientar la intervención médica y evitar la extrapolación del conocimiento obtenido en poblaciones de origen europeo (Popejoy y Mc Fullerton, 2016; Dopazo, Llera, Berenstein y González-José, 2019; Sirugo, Williams y Tishkoff, 2019).
Materiales y métodos
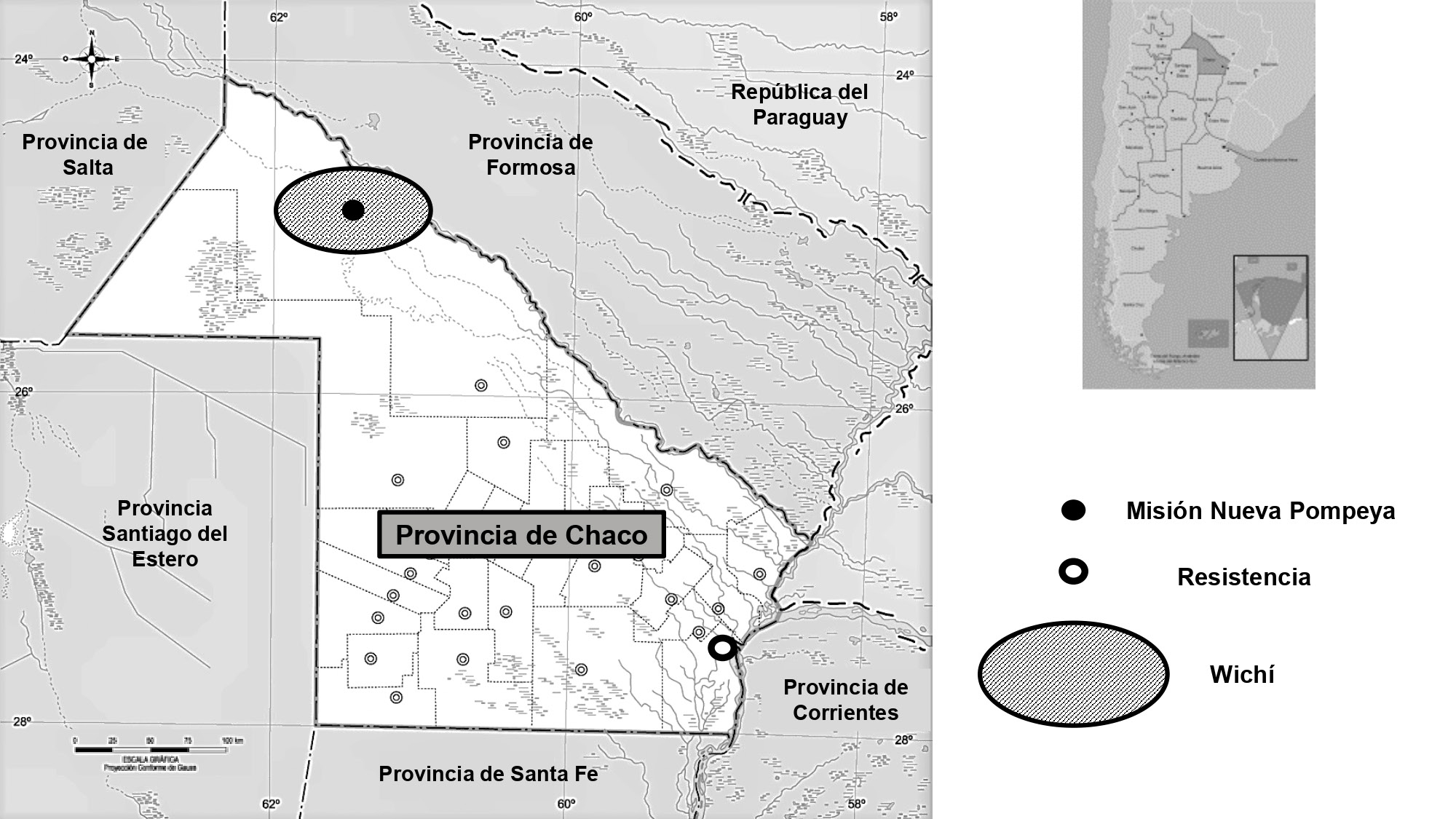
Se obtuvo ADN genómico a partir de muestras de enjuague bucal de 54 individuos habitantes de la localidad de Misión Nueva Pompeya, en el Impenetrable chaqueño (Figura 1). Los participantes voluntarios son pacientes ambulatorios adultos de ambos sexos (23 de sexo femenino y 31 de sexo masculino) que asistieron al hospital público de esa localidad durante el año 2013, cuando fueron convocados para el presente estudio. A fin de realizar comparaciones entre poblaciones chaqueñas, se incluyeron en el análisis datos previamente publicados de individuos de la comunidad wichí (n=27), también habitantes del Impenetrable, y de individuos provenientes de la ciudad capital, Resistencia (n=109) (Raggio et al., 2018).
Figura 1
Ubicación geográfica de la población de Misión Nueva Pompeya y de las dos poblaciones consideradas para su comparación en este estudio.
El proyecto fue previamente aprobado por el Comité de Ética del Instituto Multidisciplinario de Biología Celular (IMBICE) y el Ministerio de Salud de la Provincia de Chaco. Todas las muestras fueron tomadas bajo consentimiento informado de los donantes.
Las extracciones de ADN se realizaron mediante un protocolo estándar (Gemmel y Akiyama, 1996) con Proteinasa K y cloruro de litio y se cuantificaron en espectrofotómetro NanoDrop 2000c UV-Vis Spectrophotometer (Thermo Scientific, USA). La Tabla 1 detalla los SNPs analizados, su ubicación (gen, locus y localización dentro de este), la variación nucleotídica descrita en cada caso y la predicción proteica (cuando existe cambio de aminoácido).
Tabla 1
Polimorfismos genéticos analizados, su localización (GRCh38.p13) y su predicción proteica
|
Gen |
Locus |
SNP |
Localización (GRCh38.p13) |
Nomenclatura de la variante según HGVS |
Predicción proteica |
|
Cromosoma |
ARNm |
|
OPRM1 |
6q25.2 |
rs1799972 |
154039561 |
NC_000006.12:g.154039561C>T |
NM_000914.5:c.17C>T |
p.(Ala6Val) |
|
rs1799971 |
154039662 |
NC_000006.12:g.154039662A>G |
NM_000914.5:c.118A>G |
p.(Asn40Asp) |
|
rs17174794 |
154089975 |
NC_000006.12:g.154089975C>G |
NM_000914.5:c.440C>G |
p.(Ser147Cys) |
|
rs2075572 |
154090869 |
NC_000006.12:g.154090869G>C |
NM_000914.5:c.644-83G>C |
p.(=) |
|
rs540825 |
154093311 |
NC_000006.12:g.154093311A>T |
NM_000914.5:c.1164+1839A>T |
p.(Gln402His) |
|
rs562859 |
154093438 |
NC_000006.12:g.154093438C>T |
NM_000914.5:c.1164+1966C>T |
p.(Leu445=) |
|
COMT |
22q11.21 |
rs740603 |
19957654 |
NC_000022.11:g.19957654A>G |
NM_000754.4:c.-91-3545A>G |
p.(=) |
|
rs6269 |
19962429 |
NC_000022.11:g.19962429A>G |
NM_000754.4:c.1-98A>G |
p.(=) |
|
rs4633 |
19962712 |
NC_000022.11:g.19962712C>T |
NM_000754.4:c.186C>T |
p.(His62=) |
|
rs4818 |
19963684 |
NC_000022.11:g.19963684C>G |
NM_000754.4:c.408C>G |
p.(Leu136=) |
|
rs4680 |
19963748 |
NC_000022.11:g.19963748G>A |
NM_000754.4:c.472G>A |
p.(Val158Met) |
La tipificación de los 11 SNPs se realizó mediante amplificación por PCR alelo-específica o PCR y digestión con enzimas de restricción (RFLP) de acuerdo con publicaciones previas (Bergen et al., 1997; Gelernter, Kranzler y Cubells, 1999; Smith et al., 2005; Beuten, Payne, Ma y Li, 2006; Diatchenko et al., 2006; Huerta et al., 2007; Vargas-Alarcón et al., 2007). La separación de los fragmentos amplificados se realizó por electroforesis en geles de agarosa al 2% o poliacrilamida al 8%, según el tamaño de los fragmentos esperados. El análisis de los datos se realizó mediante el programa Arlequin v.3.5 (Excoffier y Lischer, 2010) para estimar el ajuste al equilibrio de Hardy-Weinberg por test exacto, y la variación entre poblaciones tomadas de a pares, por índice Fst de Wright.
Resultados
En las Tablas 2a y 2b se presentan respectivamente las frecuencias genotípicas y alélicas observadas para cada uno de los once SNPs estudiados. Las frecuencias genotípicas halladas en ambos genes se ajustaron al equilibrio de Hardy-Weinberg (p>0,05) para la muestra de la población de Misión Nueva Pompeya (MNP), salvo el SNP rs17174794 de OPRM1, que resultó monomórfico.
Tabla 2a
Frecuencias genotípicas (a) y alélicas (b) de los SNPs analizados en las poblaciones de Misión Nueva Pompeya MNP (el presente trabajo), Resistencia y Wichí (Raggio et al., 2018). Tamaños muestrales para MNP: COMT n=54; OPRM1 n=30.
|
Gen |
SNP |
Genotipo |
MNP |
Resistencia |
Wichí |
|
OPRM1 |
rs1799972 |
CC |
0,967 |
0.984 |
1 |
|
CT |
0,033 |
0.016 |
0 |
|
TT |
0 |
0 |
0 |
|
rs1799971 |
AA |
0,467 |
0.606 |
0.769 |
|
AG |
0,533 |
0.345 |
0.192 |
|
GG |
0 |
0.049 |
0.039 |
|
rs17174794 |
CC |
1 |
0.976 |
1 |
|
CG |
0 |
0.024 |
0 |
|
GG |
0 |
0 |
0 |
|
rs2075572 |
CC |
0,276 |
0.160 |
0.269 |
|
CG |
0,586 |
0.480 |
0.539 |
|
GG |
0,138 |
0.360 |
0.192 |
|
rs540825 |
TT |
0,483 |
0.550 |
0.769 |
|
TA |
0,448 |
0.383 |
0.154 |
|
AA |
0,069 |
0.067 |
0.077 |
|
rs562859 |
TT |
0,37 |
0.556 |
0.615 |
|
TC |
0,444 |
0.379 |
0.308 |
|
CC |
0,185 |
0.065 |
0.077 |
|
COMT |
rs740603 |
AA |
0,389 |
0.198 |
0.864 |
|
AG |
0,556 |
0.528 |
0.136 |
|
GG |
0,056 |
0.274 |
0 |
|
rs6269 |
AA |
0,531 |
0.417 |
0.769 |
|
AG |
0,449 |
0.444 |
0.231 |
|
GG |
0,02 |
0.139 |
0 |
|
rs4633 |
CC |
0,269 |
0.278 |
0,185 |
|
TC |
0,596 |
0.583 |
0.482 |
|
TT |
0,135 |
0.139 |
0.333 |
|
rs4818 |
CC |
0 |
0.111 |
0 |
|
GC |
0,404 |
0.407 |
0.160 |
|
GG |
0,596 |
0.482 |
0.840 |
|
rs4680 |
AA |
0,102 |
0.148 |
0.154 |
|
AG |
0,633 |
0.537 |
0.500 |
|
GG |
0,265 |
0.315 |
0.346 |
Tabla 2b
Frecuencias genotípicas (a) y alélicas (b) de los SNPs analizados en las poblaciones de Misión Nueva Pompeya MNP (el presente trabajo), Resistencia y Wichí (Raggio et al., 2018). Tamaños muestrales para MNP: COMT n=54; OPRM1 n=30.
|
Gen |
SNP |
Alelo |
MNP |
Resistencia |
Wichí |
|
OPRM1 |
rs1799972 |
C |
0,983 |
1,000 |
1,000 |
|
T |
0,017 |
0,000 |
0,000 |
|
rs1799971 |
A |
0,733 |
0,750 |
0,865 |
|
G |
0,267 |
0,250 |
0,135 |
|
rs17174794 |
C |
1,000 |
0,986 |
1,000 |
|
G |
0,000 |
0,014 |
0,000 |
|
rs2075572 |
C |
0,569 |
0,368 |
0,462 |
|
G |
0,431 |
0,632 |
0,538 |
|
rs540825 |
A |
0,293 |
0,243 |
0,154 |
|
T |
0,707 |
0,757 |
0,846 |
|
rs562859 |
C |
0,407 |
0,250 |
0,231 |
|
T |
0,593 |
0,750 |
0,769 |
|
COMT |
rs740603 |
A |
0,667 |
0,462 |
0,932 |
|
G |
0,333 |
0,538 |
0,068 |
|
rs6269 |
A |
0,755 |
0,634 |
0,885 |
|
G |
0,245 |
0,366 |
0,115 |
|
rs4633 |
C |
0,567 |
0,569 |
0,574 |
|
T |
0,433 |
0,431 |
0,426 |
|
rs4818 |
C |
0,202 |
0,315 |
0,080 |
|
G |
0,798 |
0,685 |
0,920 |
|
rs4680 |
A |
0,418 |
0,417 |
0,404 |
|
G |
0,582 |
0,583 |
0,596 |
La Tabla 3 muestra la comparación entre poblaciones tomadas de a pares mediante índice Fst. Todas las comparaciones resultaron significativas para un nivel de confianza del 95% (p<0,05), con el mayor valor de diferenciación en la comparación de la población de Resistencia con la nativa wichí (p<0,001).
Tabla 3
Porcentajes de diferenciación entre poblaciones tomadas de a pares (índice Fst/valores de probabilidad). Todos los valores resultaron significativos (p<0,05).
|
|
Misión Nueva Pompeya |
Resistencia |
|
Resistencia |
1,507%/ p=0,03604 ±0,0148 |
* |
|
Wichí |
2,145%/ p=0,04505±0,0152 |
2,744%/ p=0,00000±0,0000 |
Discusión
En este trabajo se presenta la variación de 11 marcadores genéticos de tipo SNP localizados en los genes OPRM1 y COMT en la población de Misión Nueva Pompeya; el conocimiento de la variación de estos genes es de particular interés por su potencial utilización en medicina paliativa personalizada.
Si bien la localidad de MNP se sitúa en un lugar de difícil acceso en el Impenetrable chaqueño (Altamirano y Salvay, 1996), los resultados obtenidos en este trabajo sugieren una considerable diversidad poblacional, con solo un hallazgo de ausencia de variación para un marcador del gen OPRM1. En un trabajo previo con marcadores genéticos de tipo STR e indel localizados en cromosoma X (Glesmann, 2015), la población de MNP mostró valores de heterocigosis observada mayores que los de heterocigosis esperada, lo cual apoyaría la existencia de miscegenación en dicha población. En este sentido, la población de esta localidad mantiene cierto flujo migratorio con otras localidades de Chaco, dado principalmente por jóvenes que viajan para llevar a cabo estudios universitarios o en busca de trabajo y suelen volver a su lugar de nacimiento, muchas veces con familias conformadas (Maeder, 2012). Se suma a este flujo un conjunto de trabajadores estatales que provienen de la capital del departamento Gral. Güemes, así como también de la capital provincial.
Cuando se compararon los datos de la población de MNP con aquellos de la población de Resistencia, por un lado, y la comunidad Wichí, por otro, en ambos casos se observaron diferencias significativas.
La población de Resistencia está formada por descendientes de criollos y europeos (principalmente italianos) y descendientes de los pueblos nativos (mayormente de la comunidad qom). Resistencia ocupa un área muy conectada con la ciudad capital de Corrientes a través del río Paraná y presenta un flujo migratorio mucho mayor que MNP, con movimientos desde la zona rural y del interior provincial hacia la capital chaqueña, que ocasiona el crecimiento de esta población urbana (De Pompert de Valenzuela, 2008). Según datos del INDEC (2010), Resistencia es la ciudad más poblada de su provincia y la segunda del noreste argentino. En consecuencia, a pesar de encontrarse en la misma provincia, la diferente accesibilidad influye de manera importante en la diferenciación de MNP con el área capitalina que, por su ubicación geográfica, es una importante zona de intercambio del noreste argentino, en contraste con el bajo flujo de personas en la localidad del Impenetrable.
Con respecto a las diferencias halladas en la comparación de MNP con la población wichí, las causas de estas observaciones serían distintas de las mencionadas en la comparación con la ciudad capital. Las comunidades wichí habitan en el Impenetrable, en muchos casos en los alrededores de la localidad de MNP, por lo tanto, la distancia no sería un factor determinante de diferenciación entre poblaciones. En su lugar, juegan un papel central las diferencias culturales y el modo de vida de estas comunidades como factor de aislamiento de la población mixta urbana, e incluso de otras comunidades nativas, lo cual lleva a evitar uniones de pareja con individuos por fuera de la propia comunidad (Franceschi y Dasso, 2010). Asimismo, la población wichí ha estado sometida a un proceso de deriva genética importante que estaría actuando aún en la actualidad y que se ve reflejado en la ausencia de algunos genotipos para tres SNPs de COMT (lo cual conduce a desajustes en el equilibrio de Hardy-Weinberg) y en la fijación de un alelo para dos SNPs de OPRM1. La pérdida de alelos y la tendencia a la fijación alélica y al monomorfismo son características de la deriva genética. Si bien el tamaño de muestra analizado para esta comunidad es reducido, los efectos de la deriva son claros, e incluso se han informado con mayor número de individuos analizados, haciéndose también extensivos a otras comunidades nativas chaqueñas (Goicoechea et al., 2001; Demarchi y Mitchel, 2004; Catanesi et al., 2007; Crosetti et al., 2008; Toscanini et al., 2008; Glesmann et al., 2013; Catanesi et al., 2015).
Cabe mencionar que la población capitalina de Resistencia y la comunidad wichí mostraron el mayor valor de diferenciación interpoblacional, con un Fst de 2,744%. Teniendo en cuenta que se trata de regiones codificantes, cuya secuencia nucleotídica es más conservada que la de regiones no codificantes, este valor resulta considerable. Dicha diferenciación encuentra sustento en distintos factores. Un trabajo previo de Di Fabio Rocca y colaboradores (2015) sobre linajes uniparentales de los resistencianos mostró un 67% de linajes mitocondriales nativos, pero no se hallaron linajes masculinos nativos, asimetría propia del mestizaje entre europeos y nativos que es característica de las poblaciones latinoamericanas (Wang et al., 2008; Hedrick, 2017; Caputo et al., 2021). Debe considerarse la gran movilidad de individuos en la población resistenciana, que comparte actividades laborales y académicas con la vecina ciudad (también capital) de Corrientes, con la cual no presenta diferencias significativas para OPRM1 (Raggio et al., 2018). Otro factor a tener en cuenta es el efecto de la deriva genética, que ha sido demostrada como proceso de cambio de fuerte influencia sobre la diversidad de las comunidades nativas chaqueñas, como se comentó más arriba.
Los estudios que involucran poblaciones latinoamericanas muestran diferenciación en la comparación de estas con otras poblaciones del mundo (Huerta et al., 2007; Vargas-Alarcón et al., 2007), pero las diferencias halladas entre poblaciones en este trabajo marcan la gran diversidad que existe en las poblaciones de la provincia de Chaco.
Hasta el momento, para la población argentina no se cuenta con suficiente información acerca de los efectos de estas diferencias de variación genética sobre la percepción del dolor y el requerimiento de analgesia. Los estudios sobre la variabilidad de los genes relacionados con la sensibilidad al dolor y la respuesta a analgésicos como OPRM1 y COMT son un paso previo fundamental para la toma de decisiones a la hora de indicar un tratamiento paliativo personalizado. La caracterización de variantes genéticas asociadas a sensibilidad al dolor y respuesta a su tratamiento es una tarea aún pendiente tanto en la población argentina como en la de países vecinos. El planteamiento de estas cuestiones ha conducido a emprender estudios regionales, como es el caso en Argentina del proyecto colaborativo PoblAr, de reciente inicio, que tiene como objetivo producir información genética de calidad y generar un banco de datos genéticos de nuestra población (Dopazo et al., 2019). La información que se obtenga promete no solo brindar tratamiento a un abanico de enfermedades, sino también contribuir a la aplicación de tratamientos analgésicos o paliativos específicos, basados en datos propios y no en aquellos obtenidos de otras poblaciones.
![]() 0000-0001-9799-1629 Diana M. Hohl[1][**]
0000-0001-9799-1629 Diana M. Hohl[1][**]
![]() 0000-0002-6532-6015 Laura A. Glesmann[2][***]
0000-0002-6532-6015 Laura A. Glesmann[2][***]
![]() 0000-0002-5970-5027 Cecilia I. Catanesi[1][****]
0000-0002-5970-5027 Cecilia I. Catanesi[1][****]