Introducción
El término o apelativo chaná, solo o formando otros gentilicios compuestos, aparece registrado en los primeros cronistas europeos que penetraron en la región litoraleña en el siglo XVI para designar a comunidades indígenas de la región, a veces diferenciados entre sí territorial y culturalmente. Se consigna el nombre simple chanás en la zona de Sancti Spiritu a través de Luis Ramírez (1941) en 1528 y de Diego García de Moguer en 1530 (Medina, 1908); y charnais en el delta, según consta Lopes de Sousa en sus diarios de los años 1530 y 1532 (Barlow, 1932; Politis, 2014), en el reparto de encomiendas de Buenos Aires realizado por Juan de Garay en 1582 (Outes, 1897) y en Entre Ríos (Cornero y Green, 2019). Los nombres compuestos chanatimbus, chanaes atembures se consignan también en la comarca de Sancti Spiritu por Ramírez (1941) y García de Moguer (Medina, 1908), chaná-timbús (Schmidel, 1903), y beguoá chaná (Oviedo y Valdés, 1547), entre otros.
En el interior de la Mesopotamia argentina se designa con un gentilicio compuesto a unos chanas salvajes por Schmidel (1903) y por Oviedo y Valdés (1547). Estos últimos fueron contactados por Schmidel, con el gentilicio de zechennaus saluaíschco, en la confluencia del arroyo Guayquiraró, hidrónimo histórico que fue asociado a este mismo pueblo por Canals Frau (1986). En las descripciones han sido claramente diferenciados de los anteriores en cuanto a la lengua, la ubicación territorial, las estrategias económicas y el aspecto físico (Schmidel, 1903). Los chanás salvajes fueron asociados a las poblaciones kaingang del sureste de Brasil por Serrano (1936 y 1955) y por Canals Frau (1986), quien además los vincula a pueblos yaros (Canals Frau, 1940).
Luego de la llegada de los conquistadores, indígenas chanás y de otras etnias fueron agrupados en las reducciones franciscanas de Santiago de Baradero, Santo Domingo Soriano y San Bartolomé de los Chanás. La primera fue fundada por Hernandarias en el año 1615 sobre la orilla occidental del río Baradero, afluente del bajo Paraná, en la provincia de Buenos Aires (Argentina). Allí se redujo inicialmente a unas 250 personas de origen chaná, charrúa y guaraní que habitaban las costas e islas del Paraná inferior. La segunda reducción, Santo Domingo Soriano, se estima que fue fundada en 1624 en la isla Vizcaíno (Uruguay) y estuvo habitada por chanás, bohanes y charrúas. En 1708, esta reducción se trasladó a su emplazamiento definitivo en la margen izquierda del río Negro, cerca de su desembocadura en el río Uruguay, unos 160 km al este de la reducción de Baradero. La tercera reducción se ubicaba 235 km más al norte, San Bartolomé de los Chanás, próxima a la actual localidad de Monje, en el departamento San Jerónimo, provincia de Santa Fe (Argentina).
Desde el sur y el centro de las actuales provincias de Santa Fe, Corrientes y Entre Ríos se produjo un proceso de mestizaje promovido por la toma de cautivos y el rapto de mujeres, así lo señalan algunos cronistas como Alvar Núñez Cabeza de Vaca (2007), y autores como Susnik (1966), Areces, López, Regis y Tarragó (1999) y Bracco (2016, 2017). En las reducciones se observa la convivencia de gente de diversos orígenes (Lockhart, 1975; Barreto Messano, 2011; Tapia, Nespolo y Noya, 2015; Bracco, 2017), y entre los grupos que deambulaban por el interior entrerriano en los siglos XVII y XVIII, donde se encontraban tolderías en las que convivían guenoas, charrúas, yaros, bohanes y chanás. Para el año 1686, encontramos un padrón de los indios de Santo Domingo Soriano: “Lorenzo Tuerto, huido, por quien se escribió. Marcos, huido en los charrúas. Todos los dichos chanáes”. Este padrón está incluido en el informe de Policarpo Duffo de 1715, sobre la expedición contra las tribus charrúas de Entre Ríos (Pérez Colman, 1936). Entre las investigaciones etnohistóricas que han contribuido al conocimiento de los procesos indígenas de la región se reconocen los trabajos de Lafone Quevedo (1897), Martínez (1917), Gandía (1932), Ravignani (1934), Serrano (1936), Pérez Colman (1936), Acosta y Lara (1955), Cervera (1908), de Fernández Díaz (1973), Zapata Gollán (1989), Areces et al. (1999), Bonomo et al. (2011), Bonomo y Latini (2012), Tapia et al. (2015), Politis (2014), Apolinaire y Bastourre (2016), Cornero y Green (2017, 2022 y 2024), entre otros.
Sobre la lengua chaná, el único antecedente conocido fue el estudio realizado por el Franciscano Dámaso Larrañaga Compendio del idioma de la nación chaná, escrito en el año 1815 luego de entrevistar a tres informantes ancianos en la reducción de Santo Domingo Soriano (Larrañaga, 1923).
Casi 200 años después, a partir del hallazgo de un hablante entrerriano, el periodista Daniel Fiorotto publicó una nota en un diario argentino sobre la sobrevivencia de la lengua chaná, considerada extinta hasta ese momento (Fiorotto, 2005). Este hablante, llamado Blas Wilfredo Omar Jaime, nació en la región central de la provincia de Entre Ríos, Argentina, en el seno de una familia autorreconocida como perteneciente al pueblo indígena chaná.
El lingüista Pedro Viegas Barros inició investigaciones sistemáticas trabajando con Blas Jaime en el rescate de más de 300 vocablos y expresiones de la lengua chaná, que correlacionó con el compendio de Larrañaga. Los estudios de Viegas Barros sobre morfosintaxis, fonética, fonología, gramática y comparaciones lingüísticas tuvieron amplia divulgación y aceptación en el mundo académico (Viegas Barros, 2006, 2008a, 2008b, 2009; Jaime y Viegas Barros, 2013), y lograron el reconocimiento de la lengua en organismos internacionales como la UNESCO.
El trabajo de Tapia et al. (2015) constituye el primer antecedente de análisis genéticos para la determinación de haplogrupos mitocondriales en individuos asignables a pueblos chaná, a partir de muestras procedentes del Cementerio Indígena de la reducción de Santiago de Baradero. Posteriormente, Cardozo y colaboradores (Cardozo, Tapia y Dejean, 2018; Cardozo et al., 2021) determinaron el haplogrupo mitocondrial de 11 muestras de individuos enterrados en dicho cementerio, que presentaban una antigüedad de entre 420 ± 25 y 230 ± 50 años antes del presente (AP), compatible con el momento en que funcionaba la reducción. Las muestras analizadas fueron asignadas a los cuatro haplogrupos americanos, y seis de ellas comparten el haplogrupo B o Hg B.
En el sitio Cerro Lutz, ubicado a unos 80 km al este de Baradero (provincia de Entre Ríos), se recuperaron diversos enterratorios prehispánicos primarios y secundarios, con cronologías de entre 976 ± 42 y 730 ± 70 años AP (Mazza, 2010). A partir de un molar en un paquete funerario recuperado de este sitio, se extrajo una muestra de ácido desoxirribonucleico (ADN) y se determinó por RFLP (Restriction Fragment Length Polymorphism) su correspondencia al Hg B (Cardozo et al., 2021).
Barreto Messano (2011) realizó la reconstrucción genealógica de la reducción de Santo Domingo Soriano a través de 317 entrevistas a pobladores de Villa Soriano, Uruguay, en las cuales recopiló información de 1476 individuos pertenecientes a 13 generaciones. Se logró así una proyección temporal retrospectiva de 310 años hasta momentos cercanos al establecimiento de la reducción en territorio uruguayo. Esta reconstrucción permitió determinar la presencia de diez linajes indígenas fundadores, es decir, que están presentes en la población desde su etapa de reducción, pertenecientes a cuatro etnias o grupos distintos, incluyendo linajes chanás con registros en la reducción desde el siglo XVII. Los análisis genéticos para la determinación del linaje paterno en un descendiente de los indígenas reducidos en Santo Domingo Soriano indican un posible haplogrupo del cromosoma Y de origen indígena (Barreto Messano, 2011, p. 125).
Sin embargo, la baja resolución de estos estudios genéticos sobre linajes uniparentales no permite hacer inferencias claras sobre el origen y la evolución de los grupos humanos. En este sentido, el secuenciamiento del genoma mitocondrial completo posibilita aumentar la resolución de los análisis filogenéticos, lo que supone un aporte a la comprensión de la historia evolutiva de diferentes grupos humanos.
El objetivo de este estudio es contribuir al conocimiento, desde una perspectiva multidisciplinaria, de los contextos de la lengua chaná a partir de la información lograda desde la reconstrucción genealógica, el secuenciamiento del genoma mitocondrial y la estimación de edad de coalescencia, de Blas Jaime, un voluntario reconocido como chaná y considerado el último hablante de esa lengua.
Materiales y métodos
Reconstrucción genealógica
Los registros genealógicos y biográficos directos fueron obtenidos a través de charlas y entrevistas con Blas Jaime y miembros de su familia, como su hija, Evangelina Jaime, y su esposa, Inés Gauna, del libro Gupuaigat de Ancat Imágenes del alma (1999), de Miguel Jaime, hermano de Blas, y a través de registros censales y parroquiales archivados por la Iglesia de Jesucristo de los Santos de los Últimos Días y disponibles en línea a través del sitio Family Search (https://www.familysearch.org/es/)
Análisis genéticos
Muestra biológica y extracción de ADN
Se realizó la extracción de sangre venosa periférica en tubo de hemograma con anticoagulante EDTA y se conservó en heladera (4-8 °C) hasta su procesamiento. Se realizó una extracción automatizada a partir de 200 µL de sangre entera, utilizando el equipo MagNA Pure (Roche), que permitió recuperar ADN total a partir de leucocitos. Se utilizó el programa de extracción para ADN genómico DNA Blood SV (Roche) y se eluyó en un volumen final de 100 µL. Para la cuantificación se empleó el kit Quant-iT PicoGreen dsDNA (Invitrogen).
Secuenciación de genoma mitocondrial
A partir del ADN extraído, se amplificó el genoma mitocondrial completo mediante el método de reacción en cadena de la polimerasa (PCR) de largo alcance en dos reacciones separadas, usando la enzima Phusion High-Fidelity DNA Polymerase (NEB). Los pares de iniciadores utilizados fueron MTL-F1 5’ AAAGCACATACCAAGGCCAC 3’ y MTL-R1 5’ TTGGCTCTCCTTGCAAAGTT 3’; MTL-F2 5’ TATCCGCCATCCCATACATT 3’ y MTL-R2 5’ AATGTTGAGCCGTAGATGCC 3’ (Russlies et al., 2019), y se obtuvieron productos de PCR de 9065 y 11170 pares de bases (pb), respectivamente, que fueron visualizados en gel de agarosa Megabase 1%. Los dos fragmentos solapantes fueron purificados, cuantificados y combinados en cantidades equimolares. Posteriormente, 40 nanogramos (ng) de ADN fueron usados para la preparación de una biblioteca de ADN mitocondrial (ADNmt), utilizando el kit Illumina Nextera XT DNA Library Preparation, de acuerdo con el protocolo del fabricante. Una vez preparada la biblioteca, los productos de PCR fueron purificados usando el kit AMPure beads XP (Beckman Coulter) y, subsecuentemente, cuantificados mediante el uso del kit Quant-iT PicoGreen dsDNA (Invitrogen). Para la evaluación de calidad, se corrió 1 microlitro (µl) de biblioteca posenriquecida en un bioanalizador Agilent Technologies 2100 utilizando el chip High Sensitivity DNA según instrucciones del fabricante, esperando obtener una distribución de fragmentos de ~200-2000 pb. Finalmente, las bibliotecas fueron secuenciadas en un equipo MiSeq (Illumina) desde ambos extremos con un kit de 2 x 150 ciclos.
Análisis bioinformático
La calidad de los datos crudos de secuencia fue analizada con el programa FastQC v0.11.5. Luego se realizó la remoción de los adaptadores y recorte de lecturas para regiones con calidad menor a 20 en escala phred-33 con el programa Trimmomatic 0.36 (Bolger, Lohse y Usadel, 2014). Una nueva revisión con FastQC de la calidad de las secuencias recortadas permitió determinar que eran óptimas para continuar con el procesamiento. Estas lecturas fueron analizadas utilizando la herramienta MtoolBox v1.1, que es una cadena de procesos (pipeline) para datos del genoma mitocondrial obtenidos por secuenciamiento de alto rendimiento o NGS (Calabrese et al., 2014). El pipeline produce un archivo de variantes contra la secuencia de referencia de Cambridge revisada o rCRS (Andrews et al., 1999), que se compara con una base de datos global de genomas mitocondriales. Esta base de datos contiene hoy 5400 haplotipos de poblaciones de todo el mundo.
Análisis de datos
La secuencia completa obtenida fue verificada y corregida manualmente a partir de los archivos BAM, usando el programa IGV (Integrative Genomics Viewer) 2.3.34 (Robinson et al., 2011). Con base en las variantes o sitios polimórficos identificados, la muestra fue asignada a un linaje particular según la presencia o ausencia de mutaciones de acuerdo con la filogenia propuesta en Phylotree mtDNA tree Build 17 (van Oven y Kayser, 2009), utilizando la aplicación HaploGrep 2 (Weissensteiner et al., 2016).
Para investigar las afinidades genéticas relativas entre la muestra obtenida en este estudio y otras poblaciones sudamericanas, se realizó un análisis comparativo a partir de dos bases de datos recopilados de la bibliografía: por un lado, 3700 mitogenomas de origen nativo americano, tanto de muestras modernas como antiguas; y por otro, más de 7000 secuencias de la Región Control del ADNmt (ver detalles en García et al., 2021).
Estimación de edad de coalescencia
La edad del ancestro común más reciente (TMRCA, por su nombre en inglés, the most recent common ancestor) de los subhaplogrupos de interés se estimó mediante del estadístico ρ (Figueiro et al., 2022). Para esto, se utilizó la tasa de sustitución del genoma mitocondrial completo de Soares et al. (2009), equivalente a una mutación cada 3624 años, y se excluyeron inserciones, deleciones y mutaciones en las posiciones 16182, 16183 y 16519. Se estableció un intervalo de confianza del 95% (IC95) sumando y restando 1,96 errores estándar de ρ.
Resultados y discusión
Análisis genealógico
La reconstrucción genealógica de Blas Jaime nos remonta a una ascendencia directa de tres generaciones, que implican al menos 200 años (Figura 1).
Figura 1
Genealogía correspondiente a Blas Wilfredo Omar Jaime.
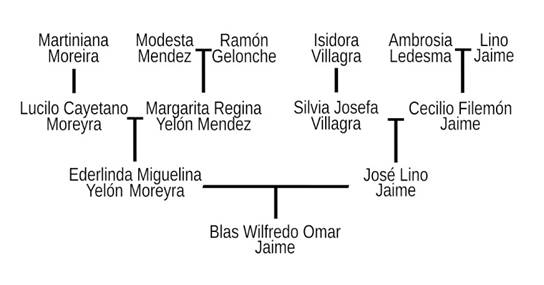
Blas Jaime (Figura 2) nació en la zona rural de Nogoyá (provincia de Entre Ríos), en el año 1934, hijo de José Lino Jaime y de Ederlinda Miguelina Yelón Moreyra (Figura 3), ambos padres reconocidos por Blas como chaná y hablantes de la lengua.
Figura 2
Blas Jaime a los 16 años en 1950, en Nogoyá, Entre Ríos.

Tanto sus abuelos paternos, Cecilio Felimon Jaime y Silvia Josefa Villagra, como los maternos, Margarita Regina Yelón Mendez y Lucilo Cayetano Moreyra, eran oriundos de la misma zona.
Figura 3
Padres de Blas Jaime. A la izquierda, Ederlinda Yelón Moreyra (1906-1998); a la derecha, José Lino Jaime (1905-1940). Cortesía de Inés Gauna.

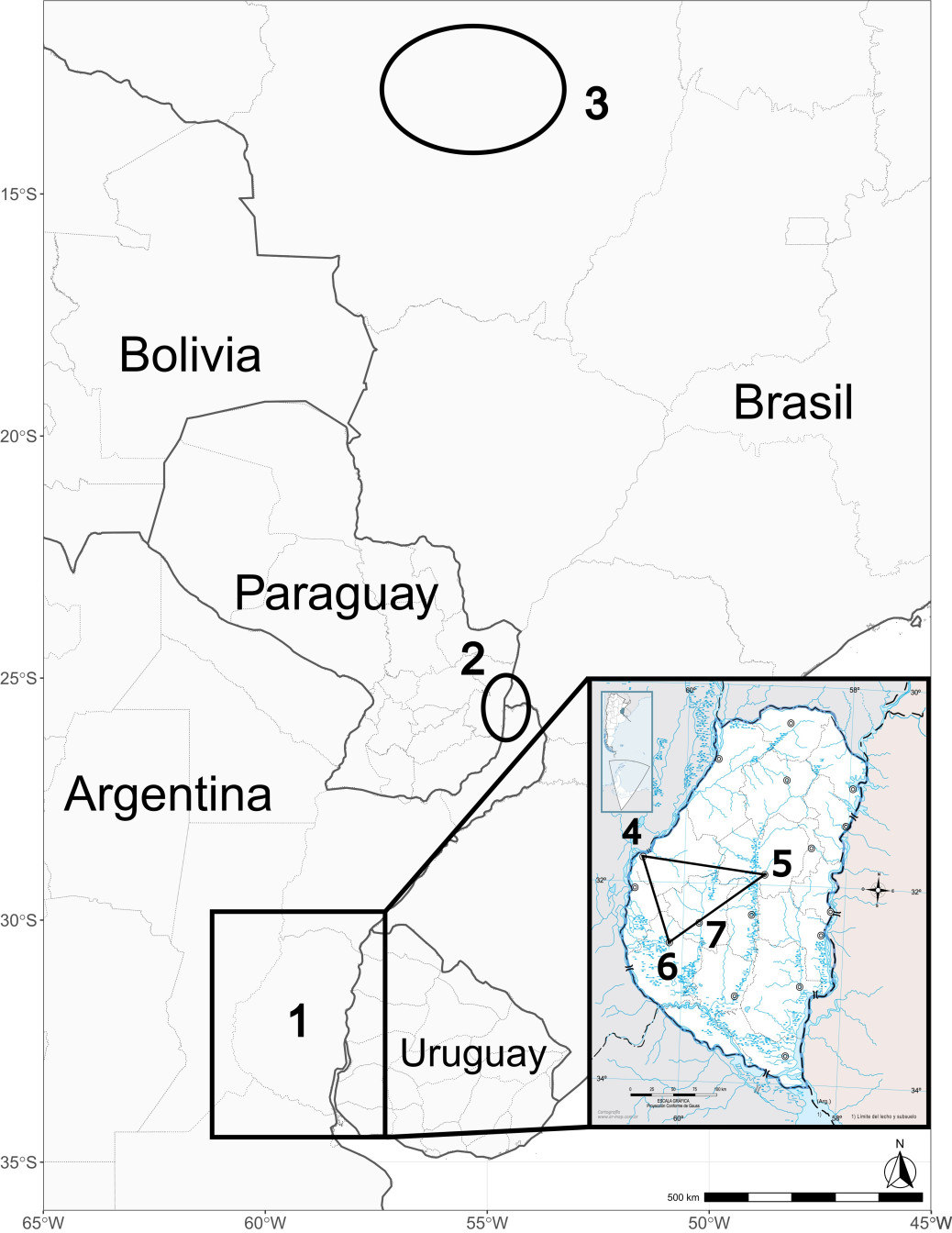
Los integrantes de estas cuatro generaciones proceden de la región central de Entre Ríos, con excepción de Martiniana P. Moreira, cuyo registro de bautismo la sitúa como oriental (uruguaya). El área aproximada de movilidad familiar se puede trazar como un triángulo entre las actuales localidades de Paraná, Villaguay y Victoria (Figura 4), con epicentro en la zona rural aledaña a Nogoyá (Estancia La Candelaria, Montes de Moreyra, y los parajes de Crucecitas, Algarrobitos y Don Cristóbal), de acuerdo con la memoria personal de Blas, su familia y a los documentos consultados, que se remontan hasta el año 1822.
Figura 4
1- Entre Ríos, área de movilidad (4- Paraná; 5- Villaguay; 6- Victoria; 7- Nogoyá.); 2- Departamento Alto Paraná, Paraguay; 3- territorios kayapó, Brasil.
Los apellidos directos que conforman el lado materno, en primer grado de consanguinidad, se reconocen y se registran en orden ascendente como Moreyra (o Moreira), Yelón (o Gelón, Gelonche) y Méndez (Mendez o Mendes), y en segundo grado de consanguinidad, Amarilla, Blanco, Berón (o Verón) y Correa. Por el lado paterno se relevaron en línea directa los apellidos Jaime (o Jaimez, Jaimes, Xaimes), Ledesma y Villagra, y en segundo grado de consanguinidad el apellido Chávez. La antigüedad de estos apellidos en la región del Río de la Plata alcanza al último tercio del siglo XVI. Algunos de ellos, como Moreyra, Méndez, Correa, Ledesma y Villagra, corresponden a vecinos encomenderos y/u ocupantes de tierras del otro lado de la banda del Paraná que llegaron con Juan de Garay desde Asunción (Paraguay) para la fundación de Santa Fe la Vieja (provincia de Santa Fe, Argentina), según consta en las Encomiendas de Buenos Aires y Santa Fe del año 1678 y el Repartimiento de Indios de Buenos Aires hecho por Juan de Garay en 1582 (Outes, 1897), y registrado por las investigaciones documentales de Calvo (1999). Los apellidos de los encomenderos eran designados a los pueblos, caciques e individuos por ellos sometidos, de modo que es esperable una relación entre apellidos europeos y familias indígenas locales. Si bien estos apellidos no se encuentran -posiblemente por subregistro- en el padrón de la reducción de San Bartolomé de los Chaná elaborado por Diego de Góngora en 1622 (Zapata Gollán, 1989), se relevan posteriormente en las reducciones de Santiago de Baradero y de Santo Domingo Soriano, desde fines del siglo XVIII a fines del siglo XIX.
Por otro lado, el apellido Amarilla, vinculado a Blas Jaime en segundo grado, es uno de los linajes fundadores chaná identificados por Barreto Messano (2011) en la reducción de Santo Domingo Soriano, junto con los del cacique Muriñigo, con registros en la reducción desde 1690, y de las hermanas Isabel y Petrona López, de las cuales se tienen registros hacia 1720. El apellido Amarilla está emparentado con Blas Jaime a partir de primos maternos, que descienden de Santiago Amarilla, cuya fecha probable de aparición en la reducción es 1730 y de quien se constituyeron ocho generaciones de varones Amarilla que llegan a la actualidad. Si bien entre los documentos de la reducción de Baradero en Entre Ríos no se registra dicho apellido, es importante destacar que las familias de filiación chaná de Baradero fueron trasladadas a Santo Domingo Soriano en 1624 durante la epidemia de viruela y tabardillo (Tapia et al., 2015; Roulet, 2019).
Análisis genético
La secuencia obtenida presenta una cobertura del 100% del genoma mitocondrial y un número promedio de lecturas por nucleótido de 1587x. Los polimorfismos identificados en la secuencia mitocondrial completa permitieron establecer que la muestra pertenece al haplogrupo B2 (Tabla 1). Sin embargo, no fue posible asignar este mitogenoma a un sublinaje conocido dentro de B2, dada la ausencia de mutaciones adicionales que sean diagnósticas de algún subhaplogrupo en la filogenia de Phylotree mtDNA tree Build 17 o en la literatura (Tabla 1).
Tabla 1
Sitios polimórficos esperados para B2 y mutaciones adicionales identificadas en la secuencia mitocondrial completa de la muestra estudiada.
|
Muestra ID |
Haplogrupo |
Sitios polimórficos esperados |
Sitios polimórficos adicionales |
|
5681112 |
B2 |
73G 263G 309.1C 499A 750G 827G 1438G 2706G 3547G 4769G 4820A 4977C 6473T 7028T 8281-8289d 8860G 9950C 11177T 11719A 13590A 14766T 15326G 15535T 16183C 16189C 16217C 16519C |
143A 956.1C 961C 16390A |
El análisis comparativo de la secuencia obtenida en este estudio reveló la ausencia de este genoma mitocondrial en la base de mitogenomas de Sudamérica. Por lo tanto, procedimos con la exploración de algún patrón de distribución espacial a partir de la búsqueda de coincidencias de cada uno de los sitos polimórficos adicionales presentes en la muestra estudiada (Tabla 1) y en la base de mitogenomas recopilados.
Por un lado, la inserción de una citosina en la posición 956 (sitio 956.1C) no fue encontrada en la base de datos y sería exclusiva de este mitogenoma. Por otro lado, la transición 961C fue hallada en seis muestras de Sudamérica (Tabla 2), correspondientes a cuatro habitantes del departamento Alto Paraná de Paraguay (Simão et al., 2019) y a dos individuos kayapó del estado de Pará, en Brasil (Fagundes et al., 2008) (Figura 4). Estas secuencias conforman el subclado B2i1, que está inmerso en el clado B2i (de Saint Pierre et al., 2012), definido por la mutación A6272G. Sin embargo, esta última mutación diagnóstica de B2i está ausente en nuestra secuencia en estudio. Dentro de B2i también está el subclado B2i2, que incluye secuencias del centro y sur de Chile y de la Patagonia argentina (de Saint Pierre et al., 2012), definido por la presencia de las mutaciones A470G-G11611A-G15077A.
Tabla 2
Muestras que poseen la mutación 961C, incluidas en los árboles filogenéticos, e información asociada
Por otro lado, realizamos un análisis comparativo basado en los sitios adicionales al haplogrupo B2 presentes en la Región Control del ADN mitocondrial de la muestra de Blas Jaime (posiciones 143 y 16390). La ventaja de una comparación a nivel de la Región Control es que existe una gran cantidad de datos disponibles de secuencias de Sudamérica y, particularmente, de la región de interés. Si bien encontramos diversas secuencias mitocondriales del haplogrupo B2 que incluyen las mutaciones 143 o 16390 en la Región Control (Bobillo et al., 2010, entre otros), el motivo completo B2+143-16390 no se encontró, hasta el momento, en ningún otro individuo de Sudamérica.
La asignación del linaje B2 a la muestra chaná de Blas Jaime está en concordancia con los resultados obtenidos en muestras poshispánicas de la reducción de Santiago de Baradero, en donde se determinó que el 55% de los individuos pertenecientes a las etnias guaraní o chaná presentaban este linaje (Cardozo et al., 2018, 2021).
En la región, una alta incidencia del haplogrupo B fue observada en poblaciones aché del este de Paraguay, en donde alcanza el 90% (Schmitt et al., 2004), mientras que este haplogrupo está ausente en grupos guaraníes, en donde predominan los haplogrupos A y D (Marrero et al., 2007). Los ayoreos del oeste de Paraguay, por su parte, presentan predominio del haplogrupo C, que llega al 80%, estando ausente el haplogrupo B (Dornelles, Bonatto, Freitas y Salzano, 2005). Entre los charrúas, el único caso conocido es el del cacique Vaimaca Perú, fallecido en el primer tercio del siglo XIX, cuyo análisis de ADNmt indica su pertenencia al subhaplogrupo C1d3, diagnosticado por la presencia de la mutación 16288C (Sans et al., 2015).
Los individuos kayapó que poseen la transición 961 provienen de dos aldeas llamadas Kriketun y Kubemkokre, correspondientes a los kayapós septentrionales de los estados de Pará y Mato Grosso, que hablan, con diferencias dialectales, una lengua perteneciente a la rama noroeste de la familia lingüística Jé, del tronco Macro-Je (Lowie, 1946; Dreyfus, 1972; Fabre, 2005). Por el contrario, los aché y los ayoreos no comparten la misma familia lingüística con los kayapós. De acuerdo con el atlas sociolingüístico (UNICEF y FUNPROEIB, 2009), los ayoreos, que viven en el área oriental de Bolivia, de transición ecocultural entre el Chaco y la Amazonía, hablan lenguas familiarizadas con el tupí-guaraní. Los aché, que viven actualmente en la región de la cordillera del Mbarakajú, en el sector oriental del Paraguay, también hablan lenguas guaraníticas. Si bien al momento se desconoce el vínculo entre la lengua chaná y el grupo Jé, Viegas Barros encuentra asociaciones con la lengua charrúa (Jaime y Viegas Barros, 2013). Carecemos de información referida a la asignación étnica de los individuos del Alto Paraná de Paraguay, ya que las muestras provienen de poblaciones urbanas (Simão et al. 2019). Sin embargo, el mapa lingüístico de Tovar (1961) ubica en esa zona a los pueblos guayaná-kaingang, opaié, y oti, incluidos en el tronco lingüístico Macro-Jé (Fabre, 2005).
Si bien el secuenciamiento del mitogenoma completo de Blas Jaime no permitió su clasificación dentro de un subhaplogrupo ya descripto de B2, la evidencia genética y el análisis de los sitios polimórficos adicionales permitieron plantear las siguientes hipótesis alternativas de relaciones filogenéticas entre este mitogenoma y otros de Sudamérica (Figura 5): a) la mutación en la posición 6272 que define B2i es revertante en el mitogenoma de Blas Jaime, por lo que esta muestra chaná estaría relacionada filogenéticamente con cuatro mitogenomas del departamento Alto Paraná de Paraguay y dos kayapós de Brasil, y lejanamente con muestras del Centro y Sur de Chile y de la Patagonia argentina; b) la mutación 6272 es un paralelismo (en este caso, B2i1 y B2i2 no estarían filogenéticamente emparentados), por lo que la muestra chaná estaría relacionada solo con los mitogenomas de Alto Paraná y kayapós a través de la mutación 961; y c) el polimorfismo en la posición 961 es un paralelismo y la secuencia chaná de Blas Jaime no está vinculada filogenéticamente con otros mitogenomas conocidos.
Figura 5
Hipótesis filogenéticas propuestas a partir de los polimorfismos observados. a) 6272 es revertante; b) paralelismo en 6272; y c) paralelismo en 961

Según la premisa básica del principio de parsimonia, los clados que comparten una mutación lo hacen porque la heredaron de un ancestro en común. Cuando este supuesto no se cumple, esa similitud se explica por eventos de reversión o paralelismo, que en conjunto se agrupan bajo el término de homoplasia. Además, el principio postula la elección de la hipótesis más probable, es decir, aquella que implique el menor número de cambios mutacionales, entre varias hipótesis concurrentes (Clark et al., 1998). Cualesquiera de las tres hipótesis propuestas en este estudio implica el mismo número de pasos mutacionales y, por lo tanto, no es factible tomar una decisión basada en el principio de parsimonia. Sin embargo, ya sea 6272 una reversión en el mitogenoma de Blas Jaime, o un paralelismo en B2i1 y B2i2, ambas opciones revelarían un vínculo filogenético con los mitogenomas kayapós y del Alto Paraná, los cuales comparten una ubicación regional en las tierras bajas sudamericanas.
Asumiendo la posibilidad de este vínculo, calculamos las edades de coalescencia de los clados B2+961 y B2i1 (Figura 5 a y b) a partir del número de diferencias entre los genomas mitocondriales de la Tabla 2 y de Blas Jaime respecto de la raíz. En el caso de B2+961, la divergencia entre el mitogenoma chaná de Blas Jaime respecto de los otros seis individuos habría ocurrido luego del surgimiento de la mutación 961 hace 12.578 (hipótesis a) o 14.566 años AP (hipótesis b), con un IC95 de 9655-15.547 y 11.398-17.787 años AP, respectivamente. Respecto del clado B2i1, se estimó que el ancestro común más reciente entre los mitogenomas kayapós y los del Alto Paraná existió hace unos 5216 años AP (IC95 entre 3523 y 6927 años).
Conclusión
A partir de la reconstrucción genealógica realizada y la evidencia histórica y documental podemos encontrar antecedentes genealógicos de Blas Jaime en las reducciones de chanás y otras etnias nativas no guaraníticas, así como en las encomiendas establecidas en el siglo XVII. Un estudio isonímico especializado podría extender los datos en el tiempo y espacio, para cubrir lagunas de información entre encomenderos, moradores y familias originarias, siguiendo como modelo el análisis genealógico de Barreto Messano (2011) en la reducción de Santo Domingo Soriano.
Por otro lado, la evidencia genética sugiere un posible vínculo filogenético entre el mitogenoma B2+961 de Blas Jaime con indígenas kayapós de los estados de Mato Grosso y Pará, en Brasil, e individuos del departamento Alto Paraná, en Paraguay. En este sentido, la región geográfica definida entre las localidades de Paraná, Villaguay y Victoria, a la cual se circunscribe la filiación histórica directa familiar de Blas Jaime desde al menos 1822, y su lugar natal, Nogoyá, quedan conectados con el departamento Alto Paraná y los territorios kayapó a través de la cuenca de los ríos Paraná-Paraguay y sus afluentes. Estos ríos han sido propuestos como antiguas rutas de menor costo para el poblamiento y desplazamiento de los grupos humanos en la región (Anderson y Gillam, 2000; Fabra, Laguens y Demarchi, 2007; Cornero, 2021). Las edades de coalescencia encontradas para B2+961 suponen un vínculo filogenético y diferenciación temprana coincidente con el poblamiento de la región, en la transición Pleistoceno-Holoceno, en concordancia con estimaciones previas realizadas para otros clados de la región (García et al., 2021; Roca-Rada et al., 2021; Motti et al., 2023).
Finalmente, el análisis de nuevos mitogenomas de poblaciones actuales y arqueológicas permitirá profundizar en el conocimiento de la diversidad mitocondrial de la región del noreste argentino, e inferir acerca de los procesos de poblamiento, y los vínculos biológicos de las distintas poblaciones entre sí y con otras de regiones geográficas de Sudamérica.
![]() 0000-0001-8137-9398 Angelina García[2][3]
0000-0001-8137-9398 Angelina García[2][3]
![]() 0000-0002-9153-0626 Rodrigo Nores[5][6]
0000-0002-9153-0626 Rodrigo Nores[5][6]